Immagine: Maurizio Pellecchia (left) and Carlo Baggio. Credit: Carrie Rosema.
Come si potrebbe impedire al nuovo coronavirus SARS-CoV-2 di entrare in una cellula ospite nel tentativo di contrastare l’infezione? Un team di scienziati biomedici ha trovato una soluzione.
Gli scienziati, guidati da Maurizio Pellecchia presso la School of Medicine dell’Università della California, a Riverside, riportano sulla rivista Molecules che due proteasi – enzimi che distruggono le proteine - situati sulla superficie delle cellule ospiti e responsabili dell’elaborazione dell’ingresso virale potrebbero essere inibite. Tale inibizione della proteasi impedirebbe al SARS-CoV2, il coronavirus responsabile di COVID-19, di invadere la cellula ospite.
Glicoproteina Spike
La superficie esterna dei coronavirus contiene una proteina critica chiamata glicoproteina spike o S-glicoproteina. Responsabile di dare al coronavirus la tipica forma a corona, la glicoproteina S è essenziale per l’ingresso di particelle virali nelle cellule ospiti. Le proteasi delle cellule ospiti, tuttavia, devono prima elaborare o tagliare questa proteina della superficie virale per consentire al virus di entrare nelle cellule.
Il laboratorio di Pellecchia e altri hanno scoperto che oltre a una proteasi precedentemente identificata chiamata TMPRSS2, il nuovo coronavirus SARS-CoV2 potrebbe anche essere elaborato da un’ulteriore proteasi umana, chiamata furina, per l’ingresso virale.
“L’uso della furina per l’ingresso virale è un meccanismo comune di entrata cellulare da parte sia delle proteine della fusione virale che di alcune tossine batteriche”, ha affermato Pellecchia, Professore di scienze biomediche, che ha guidato il gruppo di ricerca. “SARS-CoV2 utilizza anche questo meccanismo. La natura della” scissione proteolitica “nella sua glicoproteina S può determinare se questo virus può essere trasmesso attraverso le specie, ad esempio da pipistrelli o cammelli all’uomo”.
Una proteina di fusione combina gli attributi di più di una proteina. La scissione proteolitica si riferisce al processo di rottura dei legami peptidici tra aminoacidi in una proteina, che si traduce in un taglio della proteina. La glicoproteina S del coronavirus contiene tre siti di scissione che le proteasi dell’ospite umano elaborano. L’esatta natura e sequenza di questi siti di scissione e le rispettive proteasi di elaborazione, possono determinare il livello di patogenicità e se il virus può attraversare le specie.
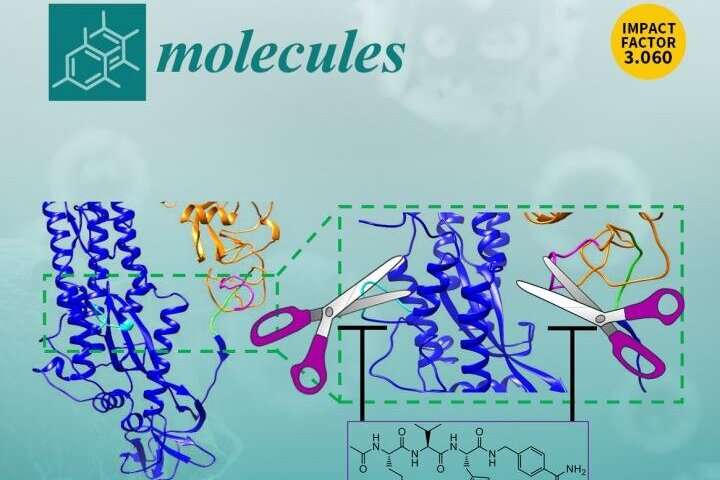
Immagine: copertina della rivista Molecules (volume 25, numero 10). Credito: Molecules, MDPI.
Il Prof. Pellecchia ha spiegato che la tossina di antrace, simile a SARS-CoV2, richiede l’elaborazione da parte della furina umana per infettare i macrofagi, un tipo di globuli bianchi. Usando la tossina antrace come sistema modello, il suo team ha scoperto che un inibitore sia della TMPRSS2 che della furina nei modelli cellulari e animali è in grado di sopprimere efficacemente l’ingresso nelle cellule dalla tossina.
Uno studio clinico con pazienti COVID-19 ha recentemente iniziato a utilizzare il Camostat inibitore TMPRSS2. “Abbiamo scoperto, tuttavia, che Camostat è un povero inibitore della furina“, ha detto Pellecchia. “Il nostro studio attuale, pertanto, richiede lo sviluppo di ulteriori inibitori della proteasi o cocktail di inibitori che possano contemporaneamente colpire sia TMPRSS2 che furina e impedire a SARS-CoV2 di entrare nella cellula ospite”.
Il Prof. Pellecchia ha aggiunto che fino ad ora la presenza di un sito di scissione della furina in SARS-CoV2 era stata collegata ad una maggiore patogenicità. Ma l’eliminazione genetica della furina negli studi di laboratorio cellulare non è riuscita a fermare l’ingresso virale, suggerendo che TMPRSS2 rimane la proteasi più rilevante.
Utilizzando sequenze di peptidi della glicoproteina S SARS-CoV2, tuttavia, il team ha ora dimostrato che le nuove mutazioni in questo ceppo di coronavirus hanno portato a un’elaborazione efficiente e aumentata dell’ingresso virale da parte di furina e TMPRSS2. “In altre parole, SARS-CoV2, a differenza di altri ceppi meno patogeni, può utilizzare in modo più efficiente entrambe le proteasi, TMPRSS2 e furina, per iniziare l’invasione delle cellule ospiti”, ha detto Pellecchia. “Mentre TMPRSS2 è più abbondante nei polmoni, la furina è espressa in altri organi, forse spiegando perché SARS-CoV2 è in grado di invadere e danneggiare più organi”.
Il laboratorio del Prof. Pellecchia ha già identificato potenti ed efficaci inibitori preclinici della furina e ha dimostrato che questi inibitori potrebbero essere sviluppati come potenziali terapie COVID-19, forse in combinazione con farmaci inibitore TMPRSS2.
Pellecchia, che detiene la cattedra Daniel Hays in Cancer Research presso la UCR School of Medicine, è stato affiancato nella ricerca da Elisa Barile, Carlo Baggio e Luca Gambini della UCR e Sergey A. Shiryaev e Alex Y. Strongin del Sanford Burnham Prebys Medical Discovery dell’ Institute La Jolla.
Fonte: Molecules


