Cancro-Immagine: i meccanismi epigenetici influenzano il modo in cui il DNA è impacchettato nei cromosomi e quindi quanto sono accessibili i geni per la trascrizione. Credito: Ella Maru Studios.
Le analisi epigenomiche suggeriscono nuovi approcci promettenti per il monitoraggio e il trattamento del cancro. Cosa stanno scoprendo le analisi e quanto sono vicine al miglioramento degli esiti dei pazienti?
Quando si tratta di cancro, i meccanismi che influenzano l’espressione dei geni senza modificarne la sequenza codificante sono importanti quanto le mutazioni genetiche. Tuttavia, la ricerca su questi processi epigenetici è rimasta indietro perché erano difficili da studiare. “Le nuove tecnologie ci stanno permettendo di recuperare il ritardo con le conoscenze genetiche”, afferma Céline Vallot, epigenetista presso l’Institut Curie di Parigi.
I processi epigenetici in genere funzionano modificando la struttura della cromatina: il DNA e le proteine istoniche che costituiscono i cromosomi. Se la cromatina è aperta e accessibile, può avvenire la trascrizione genica; dove è più compatta, i geni sono silenziati. La conformazione della cromatina può essere alterata da enzimi che etichettano chimicamente il DNA o gli istoni che avvolge. La maggior parte della ricerca epigenetica si è concentrata sui processi che influenzano questi enzimi e su come potrebbero attivare gli oncogeni o disattivare i geni oncosoppressori.
La struttura della cromatina, e quindi l’espressione genica, è influenzata anche dal DNA non codificante, che costituisce il 98% del genoma. In particolare, ci sono brevi regioni che governano l’espressione dei geni vicini e l’attenzione si sta rivolgendo alle mutazioni che influenzano questi cosiddetti potenziatori e isolanti. “Le alterazioni epigenetiche che cambiano i confini tra i quartieri genici fanno sì che i geni vengano stimolati in modo inappropriato“, afferma Howard Chang, Direttore del Center for Personal Dynamic Regulomes presso la Stanford University, in California. Il gruppo di Chang sta sperimentando metodi per studiare l’epigenoma e identificare i meccanismi chiave che regolano la trascrizione nei campi del cancro, dello sviluppo e dell’invecchiamento.
I profili trascrizionali aberranti che derivano da alterazioni epigenetiche possono essere utilizzati non solo per distinguere diversi sottotipi di cancro, ma anche come un modo per rilevare i tumori dai campioni di sangue. I farmaci che prendono di mira i regolatori epigenetici sono già stati approvati per il trattamento della sindrome mielodisplastica, dei linfomi a cellule T e del mieloma multiplo.
Tuttavia, le terapie epigenetiche devono ancora realizzare il loro pieno potenziale. Le attuali terapie sono limitate dalla tossicità e dagli effetti fuori bersaglio. I progressi nelle tecnologie di sequenziamento di nuova generazione stanno consentendo ai ricercatori di mappare le informazioni epigenomiche e i suoi effetti sulla struttura della cromatina in modo più dettagliato che mai. Studiando il panorama epigenetico delle cellule tumorali e le cellule immunitarie con cui combattono, i ricercatori stanno identificando nuovi bersagli epigenetici e approcci terapeutici. Questa conoscenza sta aiutando sia a migliorare la selettività delle terapie epigenetiche sia ad adattare i trattamenti alle caratteristiche di ciascun paziente.
Epigenetica e immuno-oncologia
L’oncologa pediatrica Crystal Mackall e il suo team della Stanford University stanno usando ATAC-seq (Assay for Transposase-Accessible Chromatin using sequencing ) per rivelare i meccanismi epigenetici che regolano l’esaurimento delle cellule T, uno stato di disfunzione progressiva che si sviluppa quando le cellule sono continuamente stimolate dagli antigeni e fattori infiammatori. Superare l’esaurimento delle cellule T è fondamentale se si vuole migliorare la durata delle terapie con cellule T del recettore dell’antigene chimerico (CAR).
“ATAC-seq fornisce una visualizzazione senza precedenti delle regioni del genoma più trascrizionalmente attive“, afferma. Il team di Mackall ha utilizzato ATAC-seq per identificare i fattori di trascrizione sovraregolati durante l’esaurimento delle cellule CAR T.
Lo scopo del team della Stanford University è interrompere i programmi trascrizionali che guidano l’espressione genica associata all’esaurimento. I ricercatori hanno trovato un probabile percorso: la sovraespressione del fattore di trascrizione c-Jun sembra prevenire l’esaurimento e migliorare il controllo del tumore, aprendo la strada a terapie CAR-T a prova di esaurimento di prossima generazione.
Combinando la conoscenza epigenetica con le tecnologie di editing genetico come CRISPR, Mackall si aspetta di vedere più “ingegneria epigenetica”, che prenderà di mira le regioni regolatrici non codificanti del genoma. “Piuttosto che rimuovere completamente l’espressione dei geni associati all’esaurimento, attraverso l’ingegneria epigenetica possiamo perfezionare la loro espressione e impedire alle cellule di reagire in un certo modo in un particolare contesto”, spiega. “Abbiamo visto che funziona, dobbiamo ancora scoprire se è utile”.
Vallot concorda sul fatto che sono necessari ulteriori miglioramenti nelle tecnologie epigenetiche prima che le scoperte attuali vengano trasmesse ai pazienti. “La tecnologia sta iniziando ad essere applicata ai campioni dei pazienti”, afferma. “Spero che, tra cinque anni, possa essere utilizzato per caratterizzare ampie coorti di pazienti per una terapia personalizzata”.
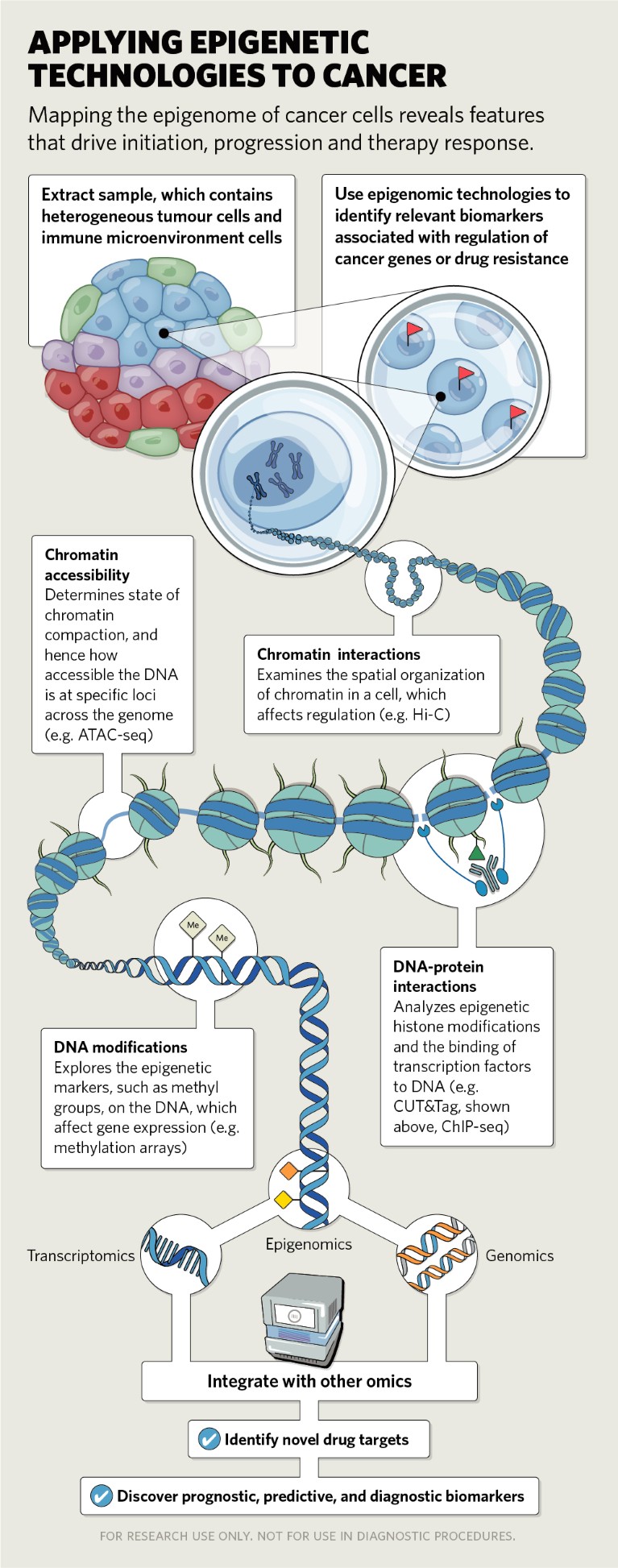
Tecnologie epigenetiche emergenti a cellula singola
Fino a poco tempo fa, la maggior parte degli studi dipendeva da tecniche che misurano gli stati epigenetici medi di popolazioni e tessuti di cellule di massa. Sebbene abbiano fornito informazioni preziose sui meccanismi generalizzati, non consentono l’esame di eventi epigenetici che si verificano in popolazioni cellulari scarsamente caratterizzate e tipi di cellule rare all’interno di tessuti normali o tumorali.
Comprendere l’eterogeneità delle cellule tumorali, sia all’interno che tra i tumori, può far luce sulle caratteristiche che determinano il potenziale metastatico delle cellule e la sensibilità al trattamento. Sono stati segnalati più di 30 metodi epigenomici a cellula singola che possono essere utilizzati per valutare la struttura della cromatina o l’accessibilità del DNA (come scATAC-seq), l’organizzazione 3-D dei cromosomi (scHI-c) e l’intero genoma modificazioni dell’istone e del DNA (scChIP-seq, scCUT&Tag) [vedi ‘Applicazione delle tecnologie epigenetiche al cancro’].
L’epigenetista dello sviluppo Melanie Eckersley-Maslin ha utilizzato tecnologie a cellula singola per studiare il rimodellamento epigenetico e l’impegno del destino cellulare durante le prime fasi dello sviluppo embrionale. Ora è entrata a far parte del Peter MacCallum Cancer Center di Melbourne, in Australia, dove il suo team applicherà questi metodi per esaminare le traiettorie del destino delle cellule tumorali.
È ben noto che le cellule tumorali possono sviluppare modelli di espressione genica che assomigliano a quelli osservati nelle cellule embrionali, ma i ricercatori sanno poco su come o perché ciò accada. “È come se le cellule avessero una crisi di mezza età”, afferma Eckersley-Maslin, “perdono la loro identità e diventano più simili a cellule embrionali, in grado di acquisire diversi destini cellulari e adattarsi a nuovi ambienti”. Gli scienziati sospettano che la comprensione del significato funzionale di questi cambiamenti possa potenzialmente rivelare nuovi biomarcatori del cancro e bersagli farmacologici.
I ricercatori dell’Institut Curie hanno sviluppato un approccio che coinvolge l’immunoprecipitazione della cromatina a cellula singola seguita dal sequenziamento (scChIP-seq) per esplorare la plasticità delle cellule del cancro al seno e le sue implicazioni per il trattamento. Utilizzando modelli di xenotrapianto di carcinoma mammario derivati da pazienti, hanno trovato un sottoinsieme di cellule sensibili ai farmaci non trattate che condividono una firma cromatinica comune con le cellule resistenti al Tamoxifene. Il loro lavoro mette in evidenza la perdita di un particolare segno cromatinico, che è associato alla repressione trascrizionale stabile (H3K27me3) come meccanismo chiave alla base della resistenza ai farmaci.
«Gli approcci a singola cellula stanno aprendo la strada alla valutazione dell’eterogeneità epigenetica all’interno dei tumori, che è fondamentale per comprendere la plasticità delle cellule tumorali e il processo di evoluzione verso la resistenza», afferma Vallot, l’autore principale dello studio. Il ricercatore si sta concentrando su come le cellule cambiano la loro predisposizione epigenetica per diventare resistenti al trattamento nel tempo e se la chemioterapia può essere combinata con modulatori epigenetici per prevenire la chemioresistenza. “Il tempismo è cruciale”, afferma. “I modulatori epigenetici devono essere forniti all’inizio della chemioterapia per limitare la plasticità delle cellule tumorali e fermare l’evoluzione della resistenza”.
Un metodo alternativo per studiare la localizzazione genomica delle modificazioni istoniche e dei fattori di trascrizione è la tecnologia Cleavage Under Targets and Tagmentation (CUT&Tag). Il materiale di partenza per CUT&Tag sono cellule permeabilizzate vive o nuclei isolati, piuttosto che cellule o tessuti reticolati con formaldeide e tagliati, come nel caso di ChIP. Con CUT&Tag, la cromatina recuperata nella fase finale del protocollo si trova nelle vicinanze locali dei siti di legame della proteina di interesse. Le sequenze di DNA più brevi isolate da CUT&Tag consentono una profondità di sequenziamento inferiore.
“CUT&Tag è un modo efficiente per profilare l’epigenoma delle singole cellule e identificare le prime fasi della malattia“, afferma il genetista specializzato in drosofila Kami Ahmad, che ha sviluppato la tecnologia con Steven Henikoff presso il Fred Hutchinson Cancer Research Center di Seattle, negli Stati Uniti. Il suo team sta applicando CUT&Tag ad alto rendimento per studiare sia la biologia del cancro negli esseri umani sia i processi di sviluppo nei moscerini della frutta. Il loro ultimo lavoro esplora il profilo epigenetico di singole cellule in campioni di pazienti con leucemia acuta a fenotipo misto. L’eterogeneità della cromatina tra le cellule dello stesso paziente potrebbe avere importanti implicazioni per il trattamento. “Sembra che le cellule possano interconvertirsi tra due stati epigenetici e che i trattamenti possano guidare il cambiamento epigenetico“, spiega.
Mettere tutto insieme
La combinazione di tecniche epigenomiche per delineare più segni epigenetici all’interno della stessa singola cellula e integrarli con informazioni trascrizionali e genetiche sarà la chiave per generare profili cellulari completi e terapie antitumorali personalizzate. “Combinando diverse tecnologie, i ricercatori stanno facendo cose davvero fantasiose e stanno ottenendo una migliore comprensione di ciò che sta accadendo nel cancro”, afferma Eckersley-Maslin.
Molti approcci multi-omici sono in fase di sviluppo per far progredire la ricerca sul cancro. “Il futuro è luminoso per la tecnologia di sequenziamento avanzata nella ricerca sul cancro”, afferma Chang.
Fonte:Nature


