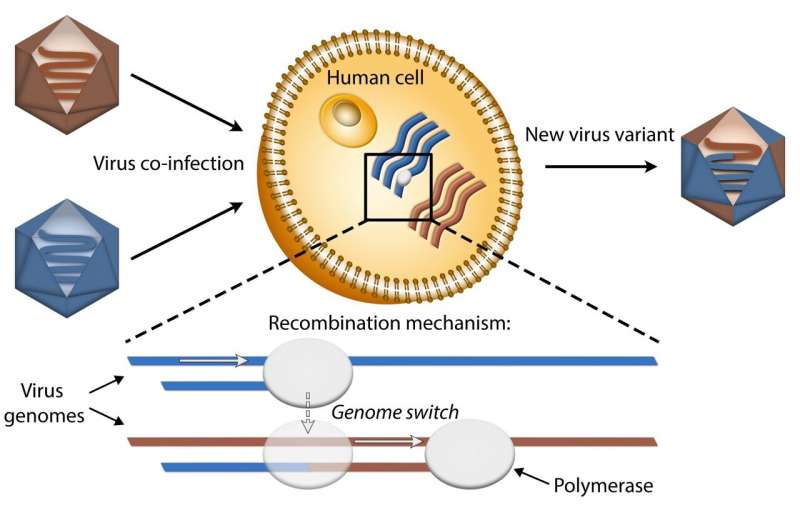
(SARS-CoV-2-Immagine: la ricombinazione virale è un importante meccanismo evolutivo nei virus a RNA che guida l’adattamento e la diversità genetica. La ricombinazione si verifica quando almeno due sottospecie di virus coinfettano la stessa cellula ospite (umana) e si scambiano segmenti genetici. Lo studio ha rivelato il meccanismo molecolare sottostante e gli inneschi della commutazione della polimerasi tra i diversi genomi del virus parentale durante la replicazione dell’RNA che può portare alla ricombinazione e alle conseguenti nuove combinazioni genetiche. Credito: TU Delft).
Un consorzio internazionale, guidato dalla Delft University of Technology e dalla University of North Carolina, è riuscito per la prima volta a sondare le origini molecolari della ricombinazione nei virus a RNA. “Il singhiozzo” durante il processo di copiatura dei virus provoca la ricombinazione: lo scambio di segmenti di RNA virale. Sebbene ciò possa portare all’emergere di nuove varianti di virus, il meccanismo può anche essere sfruttato per indurre lo sviluppo di virus difettosi non vitali.
La ricerca è stata pubblicata oggi su Molecular Cell.
La ricerca, guidata dal gruppo di ricerca del Professor Nynke Dekker, chiarisce a livello molecolare come si ricombinano i virus a RNA: quando c’è un singhiozzo o esitazione nel processo di copiatura dell’RNA (noto anche come replicazione), il meccanismo di copiatura molecolare (noto come polimerasi) salta da un filamento di RNA all’altro e continua a copiare lì. Questo provoca la ricombinazione, in cui il filamento di RNA prodotto è una combinazione di diversi filamenti di RNA, un processo che può portare allo sviluppo di nuove varianti del virus. Dekker afferma: “Abbiamo visto per la prima volta “il singhiozzo” nel processo di copiatura a livello molecolare in laboratorio, e abbiamo poi potuto osservare le sue conseguenze per la ricombinazione nelle cellule e infine nei topi. È davvero fantastico che grazie alle collaborazioni con altri istituti siamo stati in grado di capire questo meccanismo”.
Vedi anche:Varianti SARS-CoV-2: spiegato l’aumento dell’infettività
Alla ricerca di inibitori del virus
Come abbiamo sperimentato durante la pandemia della coronavirus SARS-CoV-2, le nuove varianti del virus possono avere conseguenze disastrose. È quindi fondamentale trovare modi non solo per fermare i virus esistenti, ma anche per stare al passo con le nuove varianti. I ricercatori hanno visto che il processo di copiatura e il verificarsi della ricombinazione possono essere influenzati da una nuova classe di farmaci antivirali che si rivolge specificamente a questo meccanismo di copiatura. Sembra esserci una considerazione importante da fare: quando vengono dispiegate grandi quantità di farmaci antivirali, la ricombinazione si verifica così spesso che i filamenti di RNA vengono tagliati troppo e non vengono prodotte nuove particelle virali vitali. Ma al contrario, quantità troppo basse di farmaci antivirali possono far sì che il processo di copiatura inneschi la ricombinazione a un livello praticabile, promuovendo potenzialmente la creazione di nuove varianti del virus”, ha spiegato il postdoc Richard Janissen.
Spiegano gli autori:
“Le RNA polimerasi virali RNA-dipendenti (RdRps) sono un bersaglio per agenti terapeutici antivirali ad ampio spettro. Recentemente, abbiamo dimostrato che l’incorporazione del trifosfato T-1106, un ribonucleotide pirazina-carbossammide, nell’RNA nascente aumenta la pausa e il backtracking da parte del poliovirus RdRp. Qui, monitorando la dinamica dell’enterovirus A-71 RdRp durante la sintesi dell’RNA mediante pinzette magnetiche, identifichiamo lo stato “backtracked” come un intermedio utilizzato dall’RdRp per la sintesi dell’RNA copy-back e la ricombinazione omologa. Saggi basati su cellule ed esperimenti di sequenziamento dell’RNA (RNA-seq) dimostrano ulteriormente che il ribonucleotide pirazina-carbossammide stimola questi processi durante l’infezione. Questi risultati suggeriscono che i ribonucleotidi pirazina-carbossammide non inducono mutagenesi letale o terminazione di catena, ma funzionano promuovendo la commutazione del modello e la formazione di genomi virali difettosi. Concludiamo che la commutazione del modello intra- e intermolecolare catalizzata da RdRp può essere indotta da ribonucleotidi pirazina-carbossammide, definendo un’ulteriore classe meccanicistica di ribonucleotidi antivirali con potenziale attività ad ampio spettro“.
Astratto grafico replicazione SARS-CoV-2

Immagine Credito Delft University of Technology e dalla University of North Carolina
Sono necessarie ulteriori ricerche sul meccanismo di replicazione di SARS-CoV-2 per stabilire che si tratta di un percorso efficace per fermare i virus. Dekker afferma che “l’enzima polimerasi, responsabile della replicazione del genoma, sembra essere in grado di saltare tra i filamenti di RNA senza trigger specifici. Gli inibitori del virus hanno un effetto su tale salto, ma anche la creazione di nuove varianti del virus è in agguato. Questa ricerca indica che dobbiamo essere vigili e osservare da vicino gli effetti molecolari degli inibitori del virus sia attuali che nuovi“.
Fonte:Molecular Cell