(SARS-CoV-2- Immagine Credit Public Domain).
SARS-CoV-2, che è l’agente eziologico della pandemia da COVID-19, è un coronavirus identificato relativamente di recente che da allora si è diffuso in tutto il mondo.
Si ritiene che SARS-CoV-2 sia di natura zoonotica, in quanto il virus probabilmente si è trasferito da un animale a un ospite umano a un certo punto, alla fine del 2019. Sebbene l’origine esatta di questo virus rimanga sconosciuta, è altamente probabile che abbia avuto origine da coronavirus di pipistrello ferro di cavallo o pangolino. Entrambi questi ceppi zoonotici di coronavirus presentano un gran numero di somiglianze con SARS-CoV-2 sotto diversi aspetti; pertanto, la ratifica di quale di questi ha inizialmente dato origine a SARS-CoV-2 rimane una sfida.
In un recente studio ( non peer-reviewed *) pubblicato sul server bioRxiv *, Peng Zhou, Zheng-Li Shi e colleghi dell’Istituto di virologia di Wuhan, con la collaborazione di scienziati dell’Università dell’Accademia cinese delle scienze in Cina, hanno identificato una nuova linea di coronavirus correlati a SARS che utilizzano i recettori dell’enzima di conversione dell’angiotensina 2 (ACE2) di pipistrello per l’ingresso nelle cellule, che è lo stesso recettore utilizzato da SARS-CoV-2 per infettare le cellule ospiti.
Vedi anche:I geni SARS-CoV-2 si integrano nel genoma umano?
RaTG13, RatG15 e altri coronavirus correlati a SARS
ACE2 è un enzima situato sulla membrana cellulare delle cellule principalmente respiratorie, cardiache ed epatiche negli animali ed è responsabile del controllo della pressione sanguigna. SARS-CoV-2 utilizza ACE2 come sito di ingresso per ospitare le cellule. Poiché l’ACE2 si trova principalmente nei polmoni e nel sistema respiratorio, SARS-CoV-2 e, successivamente COVID-19, spesso causano l’insorgenza di sintomi respiratori a seguito dell’infezione.
Guo e colleghi hanno campionato i pipistrelli catturati nella provincia dello Yunnan nel 2015 e hanno prelevato tamponi anali per estrarre l’acido ribonucleico virale (RNA), che sono stati poi conservati e sequenziati. Tra i virus trovati all’interno di questi pipistrelli, il team ha identificato un ceppo virale, RaTG13, come altamente simile a SARS-CoV-2, condividendo un’identità genomica del 96,2%.
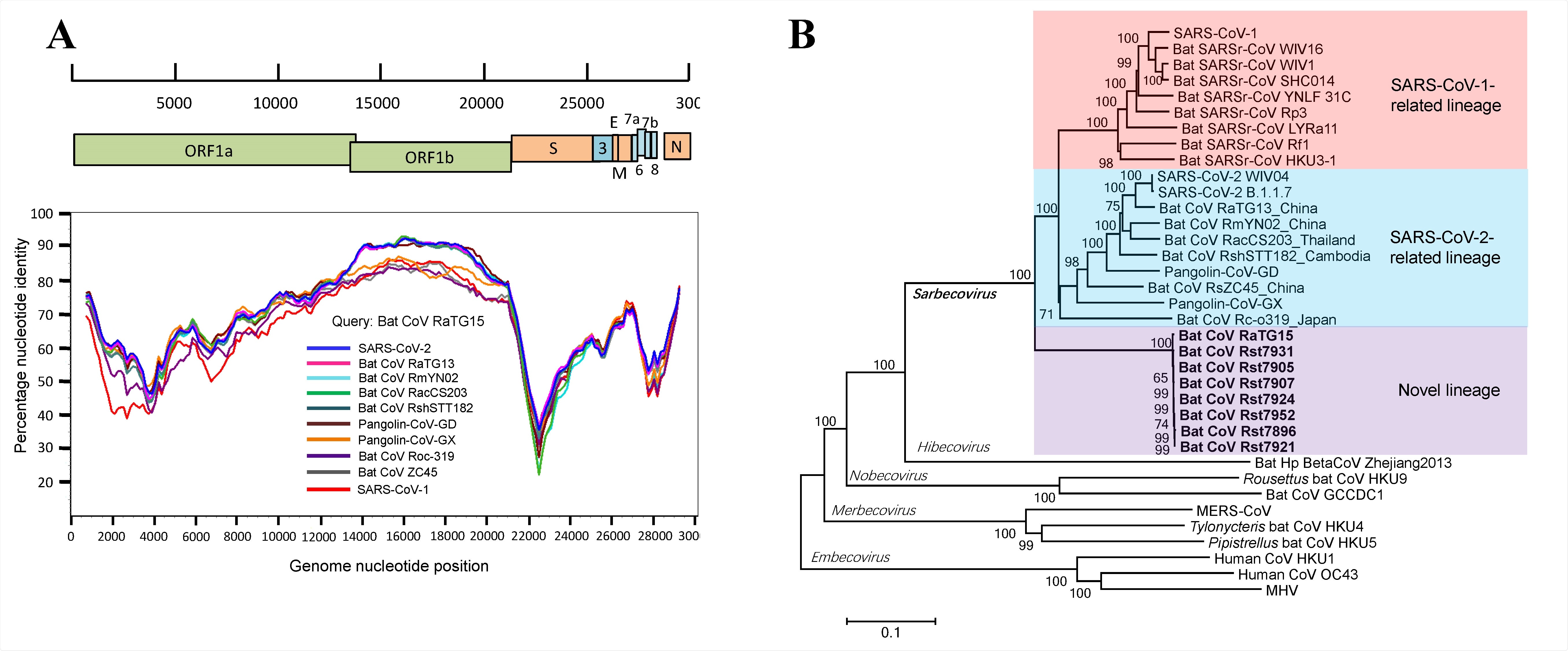
È stato inoltre riscontrato che otto ulteriori coronavirus correlati a SARS condividono il 93,5% della loro identità genomica con SARS-CoV-2. Questi otto virus erano tutti quasi identici, poiché condividevano tra loro più del 99,7% della loro identità genomica.
Uno di questi ceppi, RaTG15, è risultato simile al 95,3% a SARS-CoV-2 e al 92,5% simile a SARS-CoV-1, che è stato l’agente eziologico dell’epidemia di SARS nel 2002-2003. Attraverso ulteriori test, il team di ricerca ha concluso che RaTG15 e i suoi ceppi correlati non sono in grado di avere il potenziale zoonotico di SARS-CoV-2. Ciò contrasta con i coronavirus del pangolino, molti dei quali hanno un potenziale di ricaduta molto elevato in termini di utilizzo dei recettori cellulari.
Conclusione
Questa nuova stirpe di coronavirus di pipistrello correlati a SARS fa luce sulle potenziali origini del virus. RaTG13 e gli altri coronavirus correlati a SARS discussi in questo articolo hanno dimostrato un’affinità di legame minima per l’ACE2 umano, indicando che è improbabile che questi virus abbiano dato origine a SARS-CoV-2. Il potenziale di infezione tra specie diverse è significativamente più alto nei coronavirus correlati al pangolino, nonostante l’elevato grado di correlazione (~ 99%) tra SARS-CoV-2 e questa nuova linea di pipistrelli-CoV.
I ricercatori di questo studio evidenziano che è necessario un campionamento longitudinale di pipistrelli e pangolini, così come di altri ospiti animali intermedi, per cogliere appieno le origini della pandemia COVID-19.
Fonte: bioRxiv