(SARS-CoV-2-Immagine Credit Public Domain).
I ricercatori negli Stati Uniti hanno fornito nuove prove a sostegno di una controversa ipotesi secondo cui i geni del coronavirus Sars-Cov-2 potrebbero convertirsi in DNA e integrarsi all’interno del genoma delle cellule infette.
Il team afferma che l’RNA virale può essere espresso come trascritti chimerici con sequenze cellulari e virali fuse. “È importante sottolineare che tali trascrizioni chimeriche vengono rilevate nei tessuti derivati dal paziente”, scrive il team del Whitehead Institute for Biomedical Research di Cambridge, Massachusetts e del National Cancer Institute di Frederick, Maryland. Rudolf Jaenisch e colleghi affermano che i risultati di questo studio possono aiutare a spiegare perché alcuni pazienti che si sono ripresi dall’infezione da SARS-CoV-2 risultano ancora positivi al virus mesi dopo.
Un problema irrisolto: i pazienti che rimangono positivi all’RNA virale SARS-CoV-2
Test positivi per SARS-CoV-2 continui o ricorrenti mediante reazione a catena della polimerasi (PCR) sono stati riportati in pazienti settimane o mesi dopo il loro recupero da COVID-19. Tuttavia, nessun virus infettivo è stato isolato o diffuso da questi pazienti e la causa della continua produzione di RNA virale rimane sconosciuta.
Come altri beta coronavirus, SARS-CoV-2 utilizza una RNA polimerasi RNA-dipendente per replicare il suo RNA genomico e per trascrivere gli RNA subgenomici. Una potenziale spiegazione per il rilevamento ricorrente di RNA virale in assenza di replicazione virale è che le copie di DNA di RNA subgenomici virali possono essere integrate nel DNA della cellula ospite tramite trascrizione inversa. “La trascrizione delle copie di DNA integrate potrebbe essere responsabile di test PCR positivi molto tempo dopo che l’infezione iniziale è stata eliminata”, scrivono Jaenisch e colleghi.
Infatti, sequenze di virus RNA non retrovirali sono state rilevate nei genomi di molti animali. Diverse integrazioni in queste sequenze mostrano segnali che sono coerenti con l’integrazione di copie di DNA di mRNA virali nella linea germinale tramite retrotrasposoni a elementi nucleari (LINE) lunghi, intervallati. L’enzima in questione è una trascrittasi inversa codificata da alcune sequenze di DNA umano note come elementi LINE-1.
Inoltre, i virus a RNA non retrovirale come il virus della coriomeningite linfocitica (LCMV) possono essere trascritti inversamente in copie di DNA da una trascrittasi inversa endogena. Gli studi hanno anche dimostrato che le copie del DNA delle sequenze virali possono integrarsi nel DNA delle cellule ospiti.
Il team ha utilizzato tre diversi approcci per indagare se l’RNA di SARS-CoV-2 può essere trascritto inversamente e integrato nel genoma delle cellule umane infette in coltura. I tre approcci utilizzati per rilevare l’RNA virale sono stati il sequenziamento DNA tramite nanopori, il sequenziamento genomico intero paired-end (consente agli utenti di sequenziare entrambe le estremità di un frammento e generare dati di sequenza allineabili e di alta qualità. Il sequenziamento paired-end facilita il rilevamento di riarrangiamenti genomici e di elementi di sequenza ripetitivi, nonché fusioni geniche e nuove trascrizioni) e il sequenziamento dell’arricchimento del sito di integrazione del DNA basato su tag Tn5.
Come riportato negli Atti della National Academy of Sciences, tutti e tre gli approcci hanno fornito la prova che l’RNA SARS-CoV-2 può essere integrato nel genoma della cellula ospite.
Copie di DNA delle sequenze SARS-CoV-2 erano presenti nel genoma e hanno dimostrato di essere integrate tramite un meccanismo di retroposizione mediato da LINE1.
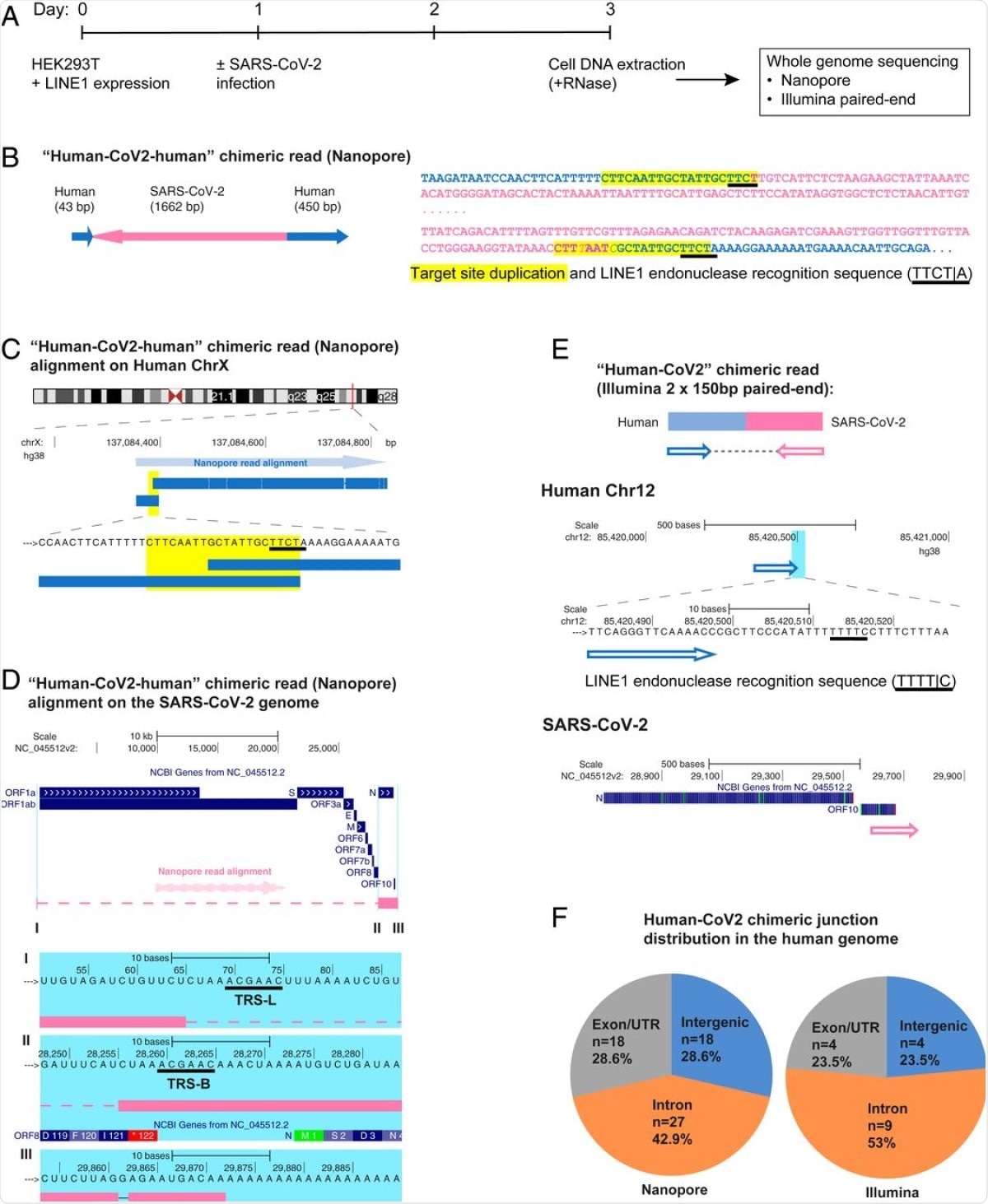
In alcuni campioni di tessuto prelevati dai pazienti, il team ha anche trovato prove che suggeriscono che una grande proporzione delle sequenze virali è stata trascritta da copie di DNA integrate di sequenze virali, generando trascrizioni chimeriche virale-ospite.
Vedi anche:“Nanotrappole per eliminare SARS-CoV-2 dai tessuti
“Questi e altri dati suggeriscono che la trascrittasi inversa LINE1 endogena può essere coinvolta nella trascrizione inversa e nell’integrazione delle sequenze SARS-CoV-2 nei genomi delle cellule infette “, scrive il team. Tuttavia, circa il 30% degli integranti virali mancava di un sito di riconoscimento dell’endonucleasi LINE1, indicando così che l’integrazione potrebbe avvenire anche tramite un altro meccanismo.
Quali sono le implicazioni dei risultati?
Jaenisch e colleghi affermano che i risultati sollevano diverse domande che richiedono ulteriori indagini. Ad esempio, i ricercatori si chiedono se le sequenze SARS-CoV-2 integrate esprimano antigeni virali nei pazienti e se queste potrebbero influenzare il decorso clinico della malattia. “Se una cellula con una sequenza SARS-CoV-2 integrata ed espressa sopravvive e presenta un virale o un neoantigene dopo che l’infezione è stata eliminata, ciò potrebbe generare una stimolazione continua dell’immunità senza produrre virus infettivo e potrebbe innescare una risposta protettiva o condizioni come l’autoimmunità come è stato osservato in alcuni pazienti ”, scrivono. Più in generale, l’integrazione del DNA virale nelle cellule somatiche può rappresentare una conseguenza dell’infezione naturale che potrebbe svolgere un ruolo negli effetti di altri comuni virus a RNA che causano malattie come la dengue e il virus dell’influenza. Questi risultati possono essere rilevanti anche per le sperimentazioni cliniche di terapie antivirali. “Se l’integrazione e l’espressione dell’RNA virale sono abbastanza comuni, fare affidamento su test PCR estremamente sensibili per determinare l’effetto dei trattamenti sulla replicazione virale e sulla carica virale potrebbe non riflettere sempre la capacità del trattamento di sopprimere completamente la replicazione virale perché i test PCR possono rilevare virali trascrizioni che derivano da sequenze di DNA virale che sono state integrate stabilmente nel genoma piuttosto che da virus infettivi “, afferma Jaenisch e colleghi.
Una parte della comunità scientifica tuttavia, si è dimostrata scettica rispetto ai risultati di qiuesto studio sostenendo che si trattasse di artefatti di laboratorio e accusando gli studiosi di alimentare timori infondati per cui i vaccini anti-Covid basati su RNA messaggero (mRNA) potrebbero in qualche modo alterare il DNA umano e al riguardo ha pubblicato un nuovo studio sulla rivista Pnas.