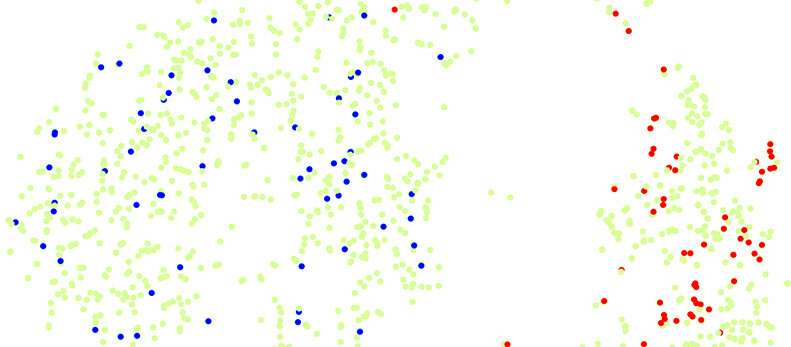
Immagine: esempio di mappatura di una popolazione eterogenea di cellule cancerose (in verde) composta da due gruppi principali. La mappatura dei picchi identifica le cellule con potenziale metastatico variabile mediante l’homing differenziale del clone metastatico (in rosso) in un gruppo e del clone non metastatico (in blu) nell’altro. Credito: © UNIGE / Ariel Ruiz i Altaba.
Prima di poter ideare un trattamento efficace, i ricercatori devono comprendere l’effetto specifico di una sostanza antitumorale sul tipo di cellula che produce metastasi nell’enorme eterogeneità cellulare dei tumori.
Un team dell’Università di Ginevra (UNIGE) ha utilizzato una tecnica pionieristica chiamata spiked-scRNAseq che collega la trascrittomica ai fenotipi metastatici monocellulari delle cellule tumorali del cancro del colon. È stata identificata l’importanza del gene VSIG1 coinvolto nelle interazioni intercellulari: il gene previene le metastasi. Oltre a questa scoperta, che offre speranza per lo sviluppo di trattamenti futuri, l’articolo pubblicato su Cell Reports convalida l’uso di questa tecnologia per testare farmaci attivi contro le metastasi, anche per approcci personalizzati.
La maggior parte dei trattamenti farmacologici per il cancro non funziona in modo ottimale contro le metastasi. Infatti, sono stati sviluppati per avere un’azione globale e il maggior effetto possibile sul paziente medio. “Per essere efficaci”, afferma il Professor Ariel Ruiz i Altaba della Facoltà di Medicina dell’UNIGE, “i farmaci dovrebbero essere mirati solo alle cellule che generano metastasi”.
I tumori sono costituiti da cellule molto eterogenee, alcune delle quali produrranno metastasi mentre altre no. La domanda è come individuare il giusto tipo di cellula tumorale come mezzo per sconfiggere il cancro.
Il team guidato dal Professor Ruiz i Altaba ha messo a punto una metodologia per definire i fenotipi cellulari per i tumori, nonché per clonarli e tracciarli, utilizzando un’analisi su scala cellulare sia del genoma che dell’espressione dell’mRNA risultante. I geni di una cellula o il suo genotipo, vengono prima copiati nell’RNA messaggero, che viene quindi spesso utilizzato per la sintesi proteica. Queste proteine sono l’espressione visibile dei geni e la loro azione è alla base delle caratteristiche misurabili della cellula, cioè il suo fenotipo. “Il nostro approccio significa che è possibile identificare i pezzi mancanti del puzzle del tumore collegando il genotipo espresso al fenotipo cellulare. In sostanza, vogliamo sapere come le cellule diventano metastatiche e da dove vengono“.
Validazione del modello
Sviluppando e utilizzando la tecnica spiked-scRNAseq, che combina approcci omici e determinazione del fenotipo di singole cellule che vengono poi “addizionate” nella popolazione cellulare eterogenea originale, i ricercatori sono riusciti a definire con precisione la composizione delle popolazioni tumorali determinando il loro fenotipo a livello cellulare.
Per fare ciò, i ricercatori hanno prima etichettato geneticamente diversi tipi di cellule tumorali in modo da poter osservare il loro comportamento metastatico. Una volta noto il fenotipo, le cellule dello stesso clone sono state “addizionate” nella popolazione di cellule primarie di cancro al colon eterogeneo e sottoposte a sequenziamento di singole cellule. Il profilo molecolare delle cellule con potenziale metastatico è stato quindi identificato dall’homing di cellule metastatiche a spillo in un gruppo o cluster cellulare specifico.
Vedi anche:Bloccare lo sviluppo delle metastasi utilizzando il riflesso di evasione
Sulla base di questo approccio, i farmaci e i loro effetti antimetastatici potrebbero essere testati con precisione. “In altre parole, possiamo usare questo approccio per analizzare i composti per l’azione sulle cellule che generano metastasi nei singoli pazienti“, spiega la Dottoressa Marianna Silvano, borsista post-dottorato presso UNIGE e co-primo autore dello studio.
Identificazione di un gene soppressore di metastasi
I ricercatori hanno quindi applicato questo approccio per comprendere il comportamento del tumore. “Abbiamo iniziato a definire lo stato pre-metastatico di un tumore e valutare il suo potenziale metastatico“, continua la Dr.ssa Silvano. Il team di ricerca di Ginevra ha scoperto che mescolando cellule che hanno un fenotipo metastatico con cellule non metastatiche, le prime hanno fermato la migrazione delle seconde. “Questo indica che le interazioni cellulari restrittive sono importanti nel processo di formazione delle metastasi”, spiega il Professor Ruiz i Altaba.
Sulla base di questi risultati – e con analisi genomiche e trascrittomiche in mano – il team di ricerca si è concentrato sui geni coinvolti nelle vie di segnalazione che sono importanti per le interazioni cellulari. La Dottoressa Carolina Bernal, borsista post-dottorato e co-autrice dello studio, ha identificato il gene VSIG1 e lei e il Dottor Silvano lo hanno scoperto come critico dell’interazione restrittiva tra cellule tumorali non metastatiche e metastatiche.
“Esprimendo il gene in una cellula tumorale, le metastasi vengono ridotte in vivo e in vitro. Se viene rimosso, troviamo un aumento delle metastasi“, conclude Silvano. Le cellule metastatiche nei tumori primari possono essere individuate con scRNAseq a spillo e geni essenziali e farmaci attivi identificati per sviluppare trattamenti nuovi ed efficaci per il colon e altri tumori.
Fonte:Medicalxpress