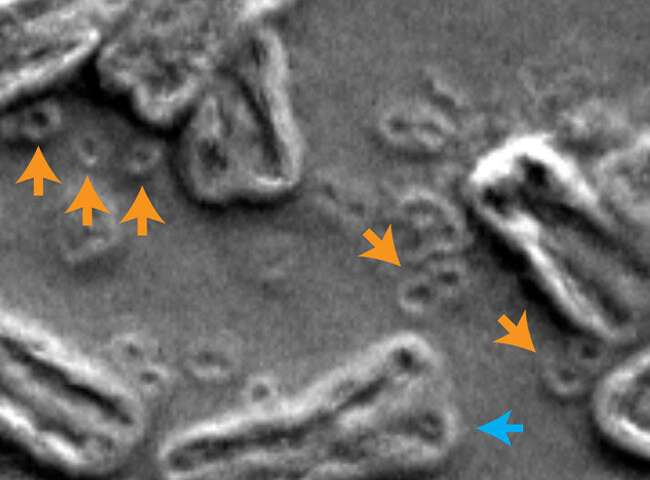
Immagine, in questa microfotografia elettronica a scansione all’interno del nucleo di una cellula cancerosa, i cromosomi sono indicati da frecce blu e DNA extra-cromosomico circolare sono indicati da frecce arancioni. Credito: Paul Mischel, UC San Diego.
L’acido desossiribonucleico o il DNA codifica le informazioni, non solo nella sua sequenza, ma anche nella sua forma. Basandosi su precedenti lavori rivelatori, un team di scienziati, guidati da ricercatori dell’Università della California di San Diego, della filiale UC San Diego del Ludwig Institute for Cancer Research, dell’Università di Stanford e dell’Howard Hughes Medical Institute, riferiscono che nel cancro, DNA extracromosomico (ecDNA) a forma di cerchi che si trovano abbondantemente nelle cellule tumorali umane, cambiano il modo in cui sono espressi gli oncogeni correlati al cancro; promuovono un comportamento maligno aggressivo e svolgono un ruolo chiave nella capacità dei tumori di evolversi rapidamente e resistere alle minacce, come la chemioterapia, le radiazioni e altri trattamenti.
“Questo studio è un cambiamento di paradigma. La forma dell’ecDNA del cancro è diversa dal normale DNA e ciò ha implicazioni davvero importanti, sia per la nostra comprensione della biologia del cancro che dell’impatto clinico”, ha affermato Paul S. Mischel, MD, Professore nell’UC Dipartimento di Patologia della San Diego School of Medicine e membro Ludwig.
Mischel è autore co-senior dello studio con Vineet Bafna, Ph.D., Professore di informatica e ingegneria presso la Jacobs School of Engineering della UC San Diego; Howard Y. Chang, MD, Ph.D., Ludwig Professore di genomica del cancro e di genetica alla Stanford University e all’Howard Hughes Medical Institute e Bing Ren, Ph.D., Professore di medicina cellulare e molecolare presso la UC San Diego School of Medicine e membro Ludwig.
Il DNA umano forma tipicamente doppie eliche lunghe e contorte di materiale genetico: circa 3 miliardi di coppie di basi organizzate in 23 coppie di cromosomi, miracolosamente schiacciate in ogni nucleo cellulare, ciascuna con una media di soli sei micrometri di diametro. (Tutto il DNA in un singolo nucleo cellulare si estenderebbe per circa sei piedi e tutto il DNA nel corpo di una persona si estenderebbe per circa il doppio del diametro del sistema solare, più di 7 miliardi di miglia.)
Nell’uomo e in altri organismi eucarioti, il DNA normale viene impacchettato nei nuclei cellulari avvolgendolo strettamente attorno a gruppi strettamente raggruppati di complessi proteici. Per accedere e leggere le istruzioni genetiche del DNA, le cellule si basano su enzimi e macchinari complicati per tagliare e spostare frammenti, rendendo accessibili solo parti alla volta.
Ma le cellule tumorali, riportano i ricercatori, condividono alcune somiglianze con i batteri, che contengono DNA circolare generalmente più accessibile. Per studiare come la forma dell’ecDNA influisce sul funzionamento, il team di ricerca ha sviluppato mappe circolari topograficamente informate del DNA extracromosomico oncogenico. Ha scoperto che l’ECDNA circolare nel cancro, sebbene avvolto attorno ai nuclei proteici, è altamente accessibile, consentendo maggiori punti di accesso e luoghi in cui le informazioni genetiche possono essere rapidamente trascritte ed espresse. Questa funzione consente alle cellule tumorali di generare enormi quantità di oncogeni che promuovono la crescita e di evolversi più rapidamente e di rispondere più energicamente al loro ambiente in evoluzione e alle potenziali minacce.
“Mostrando che l’ecDNA è circolare, quindi chiarendo la sua organizzazione epigenetica, dimostriamo qualcosa di molto potente”, ha detto Mischel. “Questa forma unica nelle cellule tumorali umane è abbastanza diversa dal normale DNA umano. Questa ricerca illumina davvero una nuova luce sull’organizzazione 3-D del genoma del cancro e dell’epigenoma, che ora fornisce una base meccanicistica per capire perché alcune cellule tumorali sono così aggressive”.
I risultati dello studio si basano su ricerche pubblicate nel 2017 che riportano che brevi frammenti di DNA circolare che codifica per i geni del cancro erano molto più comuni di quanto si pensasse in precedenza – rilevati in quasi la metà dei tumori umani, ma raramente nelle cellule normali – e probabilmente giocheranno un ruolo chiave nell’evoluzione dei tumori e nella resistenza alle minacce, come la chemioterapia.
Si pensava che il DNA extracromosomico fosse raro, ma in un articolo del 2014 Mischel e colleghi hanno scoperto che l’ECDNA svolge un ruolo centrale nella resistenza ai farmaci di alcuni tumori cerebrali consentendo ai tumori di cambiare rapidamente la quantità di oncogeni che contengono e determinare se un la cellula si trasforma in una cellula tumorale. La scoperta ha sorpreso i biologi che da tempo si sono concentrati maggiormente su quali geni promuovono il cancro piuttosto che su dove si trovano quei geni.
Le ultime scoperte sottolineano drammaticamente come le cellule tumorali non giocano secondo le stesse regole biologiche delle cellule eucariotiche.
Piuttosto che passare il DNA alle generazioni successive dividendosi in cellule figlie geneticamente identiche — un processo chiamato mitosi, che coinvolge cromosomi accoppiati che si dividono e utilizzati da tutti gli eucarioti — i batteri e il cancro si propagano distribuendo l’ecDNA alle cellule figlie in modo apparentemente casuale, fornendo un meccanismo mediante il quale alcune cellule figlie potrebbero ricevere più copie cancerose all’interno di una divisione cellulare. È un processo di ereditarietà nettamente diverso che consente una più rapida evoluzione e cambiamenti genetici.
Nell’ultimo articolo, il team di ricerca ha impiegato una serie di tecnologie avanzate: microscopia elettronica ultrastrutturale, mappatura ottica a lungo raggio e analisi computazionale del sequenziamento dell’intero genoma.
“Abbiamo utilizzato una serie di strumenti personalizzati per l’analisi delle sequenze, tra cui Amplicon Architect e Amplicon Reconstructor che ricostruiscono la struttura dell’ECDNA usando il sequenziamento dell’intero genoma e le mappe ottiche e hanno usato queste strutture ricostruite come un framework per stratificare i dati epigenetici e analizzare le differenze tra intra ed extra -Drom cromosomico “, ha detto Bafna.
Fonte, Nature