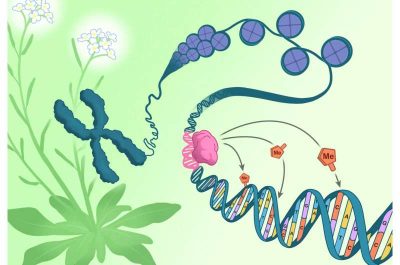
Epigenetica-immagine: un cromosoma estratto dai fiori di Arabidopsis thaliana (verde e bianco) si srotola per rivelare il DNA (blu) avvolto attorno a proteine di impacchettamento chiamate istoni (viola). La direzione dei cambiamenti epigenetici attraverso le caratteristiche genetiche inizia quando il fattore di trascrizione RIM (rosa) si aggancia a una sequenza di DNA corrispondente (rosa). Una volta agganciato, il fattore di trascrizione RIM dirige il meccanismo di metilazione per attaccare i gruppi metilici (arancione) a specifiche citosine vicine (arancione). Credito: Salk Institute.
Tutte le cellule di un organismo hanno esattamente la stessa sequenza genetica. Ciò che differisce tra i diversi tipi cellulari è la loro epigenetica, ovvero etichette chimiche meticolosamente posizionate che influenzano quali geni vengono espressi in ciascuna cellula. Errori o carenze nella regolazione epigenetica possono portare a gravi difetti dello sviluppo sia nelle piante che negli animali. Questo solleva una domanda sconcertante: se i cambiamenti epigenetici regolano la nostra genetica, cosa li regola?
Gli scienziati del Salk Institute hanno ora utilizzato cellule vegetali per scoprire che un tipo di tag epigenetico, chiamato metilazione del DNA, può essere regolato da meccanismi genetici. Questa nuova modalità di target della metilazione del DNA vegetale utilizza sequenze di DNA specifiche per indicare al meccanismo di metilazione dove agganciarsi. Prima di questo studio, gli scienziati avevano compreso solo come la metilazione del DNA fosse regolata da altre caratteristiche epigenetiche, quindi la scoperta che le caratteristiche genetiche possono anche guidare i modelli di metilazione del DNA rappresenta un importante cambiamento di paradigma.
Questi risultati potrebbero fornire informazioni per future strategie di ingegneria epigenetica volte a generare modelli di metilazione che possano riparare o migliorare la funzione cellulare, con numerose potenziali applicazioni in medicina e agricoltura.
“Nelle piante e negli animali, modelli errati di metilazione del DNA possono causare difetti dello sviluppo e, nei mammiferi, possono portare a numerose malattie, tra cui il cancro“, afferma l’autrice senior Julie Law, Ph.D., Biochimica e Prof.ssa associata al Salk Institute. “Questo rende molto importante per noi capire come la metilazione del DNA venga indirizzata nelle posizioni corrette, nei tessuti e nelle fasi di sviluppo corrette. Il nostro lavoro risponde a una domanda di vecchia data su come si generano nuovi modelli di metilazione durante lo sviluppo delle piante, che rappresenta il primo passo per pensare all’ingegnerizzazione dei modelli di metilazione del DNA per migliorare l’idoneità cellulare“.
Lo studio è stato pubblicato su Nature Cell Biology il 21 novembre 2025.
Che cos’è l’epigenetica?
Le istruzioni cellulari sono scritte in un linguaggio di quattro lettere – A, T, C e G – che si uniscono per formare lunghi filamenti di DNA. Questi lunghi e indisciplinati tratti di DNA vengono poi avvolti attorno a proteine chiamate istoni e impacchettati nella cromatina, condensando e organizzando i filamenti per facilitarne la conservazione e l’accesso. L’epigenoma è uno strato di tag e modifiche apportate sopra tutto ciò. Questi cambiamenti determinano quali geni vengono espressi e quali no senza alterare il codice base stesso, consentendo flessibilità nell’identità e nel comportamento cellulare.
“Un importante tag epigenetico è la metilazione del DNA, in cui un gruppo metilico viene aggiunto a specifiche lettere “C” all’interno del codice del DNA. Questi tag di metilazione del DNA segnalano che il DNA sottostante deve essere “disattivato”, un processo chiamato “silenziamento“. Questo processo è importante non solo per regolare l’espressione genica, ma anche per silenziare l’espressione di elementi genetici speciali, chiamati trasposoni. Se espressi, i trasposoni possono muoversi all’interno del genoma, causando instabilità genomica e riduzione dell’idoneità dell’organismo”.
Comprendere come, quando e perché in ogni tipo di cellula vengono generati specifici modelli di metilazione del DNA è fondamentale per spiegare lo sviluppo biologico e curare le malattie che comportano disfunzioni epigenetiche.
“Abbiamo imparato molto su come un tag epigenetico possa essere mantenuto dopo essere stato stabilito”, spiega Law. “Ma la diversità cellulare non deriva da modelli consolidati; deriva da modelli nuovi, e c’è ancora molto che non sappiamo su cosa crei un nuovo modello epigenetico. Questo lavoro sta colmando quel divario tra la conoscenza dell’esistenza della diversità epigenetica e la comprensione di come viene generata“.
Perché studiare l’epigenetica nelle piante?
L‘Arabidopsis thaliana è una piccola pianta infestante che per decenni è stata la pianta di riferimento per i laboratori. L’Arabidopsis tollera meglio le alterazioni sperimentali nelle modificazioni epigenetiche rispetto alle cellule umane o di altri animali, quindi è un’ottima risorsa per indagare questioni fondamentali sull’epigenetica.
In Arabidopsis, i modelli di metilazione del DNA sono regolati da una famiglia di quattro proteine chiamate CLASSY. Ogni CLASSY è responsabile del reclutamento del meccanismo di metilazione del DNA in diverse posizioni all’interno del genoma. Ma prima di questo studio di Salk, gli scienziati non avevano chiaro come CLASSY3 mediasse questo targeting. Cosa la spingeva a scegliere un set di bersagli genomici rispetto ad altri?
Come hanno inizio i cambiamenti epigenetici?
Fino a quel momento, gli scienziati avevano osservato solo che gli eventi di metilazione del DNA erano influenzati da altre caratteristiche epigenetiche. Ad esempio, se una sezione di DNA era già stata metilata per sopprimere l’espressione genica in quella regione, gli scienziati avevano capito come questa metilazione potesse essere ristabilita nella stessa posizione dopo la divisione cellulare.
Questi meccanismi di auto-rinforzo sono particolarmente importanti per il mantenimento dei modelli epigenetici durante la vita di un organismo. Ad esempio, quando una cellula cutanea invecchiata si divide in due nuove cellule cutanee, non si vorrebbe che emergesse un modello epigenetico completamente nuovo che improvvisamente riprogrammasse quelle cellule cutanee in cellule tumorali.
Ma cosa succede nei casi in cui si desidera che il modello epigenetico cambi, come durante lo sviluppo o in risposta a uno stress ambientale? In che modo una cellula vegetale modifica la sua epigenetica per crescere, rispondere e riprendersi?
“Come hanno origine questi modelli?” si chiede il primo autore Guanghui Xu, Ph.D., ricercatore post-dottorato nel laboratorio di Law. “Volevamo scoprire cosa regolasse i percorsi epigenetici per creare nuovi modelli di metilazione del DNA durante lo sviluppo, la rigenerazione e la riproduzione delle piante”.
Leggi anche:La riprogrammazione epigenetica potrebbe invertire l invecchiamento?
Un cambio di paradigma nella metilazione del DNA vegetale
Per indagare l’origine di questi modelli di metilazione del DNA, i ricercatori hanno esaminato i tessuti riproduttivi di Arabidopsis. Utilizzando uno screening genetico diretto, hanno scoperto una nuova modalità di target della metilazione del DNA che si basa sulle sequenze di DNA piuttosto che sulle caratteristiche epigenetiche.
Si è scoperto che diverse proteine, che il team ha chiamato “RIM”, interagiscono con CLASSY3 per stabilire la metilazione del DNA in corrispondenza di specifici bersagli genomici nei tessuti riproduttivi delle piante. Queste RIM sono un sottoinsieme di una vasta classe di proteine chiamate fattori di trascrizione del MERISTEM RIPRODUTTIVO (REM). Questa è stata una scoperta sorprendente, poiché ha collegato il target di CLASSY3 a specifiche sequenze di DNA. Quando gli scienziati hanno interrotto questi tratti di DNA, l‘intero percorso di metilazione è fallito.
Lo studio identifica tratti di DNA indispensabili in cui i RIM si ancorano, dopodiché possono indirizzare il meccanismo di metilazione del DNA per influenzare le sequenze di DNA adiacenti. Come risultato di questa attività di targeting, i ricercatori hanno dimostrato che nei tessuti riproduttivi che esprimono diverse combinazioni di RIM si generano modelli unici di metilazione. Questa è la prima volta che gli scienziati hanno identificato una sequenza genetica in grado di guidare il processo epigenetico di metilazione del DNA nelle piante. Poiché in Arabidopsis sono presenti molti geni REM, il team prevede che ulteriori membri della famiglia saranno collegati alla metilazione del DNA, ampliando il loro ruolo nel controllo della regolazione epigenetica.
Un altro studio pubblicato su Nature Cell Biology e condotto dal Dott. Steven Jacobsen dell’Università della California a Los Angeles ha utilizzato la genetica inversa per identificare diversi geni REM coinvolti nella regolazione della metilazione del DNA attraverso sequenze specifiche di DNA, supportando ulteriormente il ruolo delle informazioni genetiche nel guidare i processi epigenetici.
“Questa scoperta rappresenta un cambiamento di paradigma nella visione del settore su come la metilazione sia regolata nelle piante“, afferma Law. “Tutti i lavori precedenti indicavano modifiche epigenetiche preesistenti come punto di partenza per il target della metilazione, il che non spiegava come potessero emergere nuovi modelli di metilazione. Ora sappiamo che anche il DNA stesso può istruire nuovi modelli di metilazione”.
Grazie a queste nuove prove che dimostrano come le caratteristiche genetiche possano guidare i cambiamenti epigenetici, i ricercatori hanno una serie di ulteriori interrogativi da esplorare, tra cui quanto sia diffusa questa nuova modalità di targeting durante lo sviluppo delle piante e come possa essere sfruttata per progettare nuovi modelli di metilazione del DNA. La capacità di utilizzare sequenze di DNA per indirizzare la metilazione ha ampie implicazioni per l’agricoltura e la salute umana, poiché consentirebbe di correggere i difetti epigenetici con un elevato grado di precisione.
Fonte: Nature Cell Biology