Un paio di studi del laboratorio di Evangelos Kiskinis, Ph.D., Professore associato presso la Divisione di Malattie Neuromuscolari e di Neuroscienze del Dipartimento di Neurologia della Northwestern University, hanno scoperto nuovi meccanismi cellulari coinvolti in due tipi di malattie genetiche di SLA.
I risultati, pubblicati su Science Advances e Cell Reports, migliorano la comprensione della SLA, una malattia neurodegenerativa progressiva che attacca i motoneuroni nel cervello e nel midollo spinale e forniscono supporto per il futuro sviluppo di terapie mirate.
Mutazioni genetiche “adescanti”
Secondo la Les Turner ALS Foundation, negli Stati Uniti circa 32.000 persone vivono attualmente con la SLA. Esistono due tipi di SLA: sporadica (non genetica) che costituisce oltre il 90% di tutti i casi di SLA, e familiare (genetica).
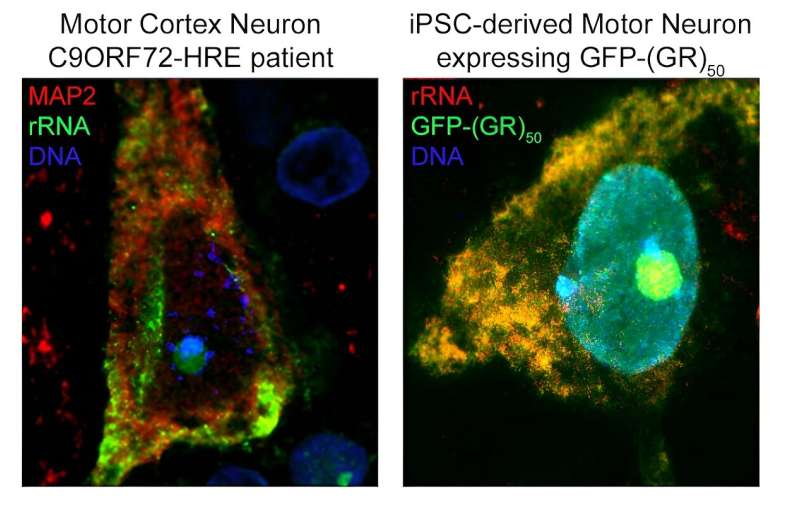
Nel loro articolo pubblicato su Science Advances, il team Kiskinis ha studiato un tipo di SLA familiare causata da una sequenza genetica ripetuta del gene C9ORF72, che è la principale causa genetica della SLA.
Secondo Kiskinis, gli individui sani possono “avere 20 ripetizioni di C9ORF72 nel loro genoma, mentre i pazienti affetti da SLA possono averne migliaia”. Queste ripetizioni vengono trascritte e tradotte attraverso percorsi non canonici che producono RNA irregolari e dipeptidi (molecole con due amminoacidi legati da un peptide) nei neuroni.
Il team di Kiskinis ha ipotizzato che “questo effetto a cascata sia ciò che porta alla tossicità da guadagno di funzione nei motoneuroni in questo tipo di SLA”. Ciò ha spinto i ricercatori a studiare i meccanismi che rendono questi dipeptidi ripetuti (R-DPR) sempre più tossici.
Utilizzando tecniche computazionali e sperimentali, i ricercatori hanno scoperto che gli R-DPR hanno una forte affinità di legame per le molecole di RNA. Quindi, hanno utilizzato una tecnica chiamata immunoprecipitazione reticolante o CLIP-seq, per isolare specifici frammenti di RNA e hanno scoperto che gli R-DPR si legano esclusivamente agli RNA ribosomiali. Nello specifico, “il dipeptide poli-GR si lega all’RNA ribosomiale, compromettendo l’omeostasi ribosomiale, un processo essenziale per la differenziazione cellulare e la composizione complessiva di una cellula”.
Utilizzando questi risultati, i ricercatori hanno quindi progettato “un’esca” di RNA che induce il poli-GR a legarsi con qualcosa che sembra essere RNA ribosomiale o, in questo caso, l’esca. Sia nei modelli in vivo che nei neuroni iPSC derivati da pazienti con mutazioni C9ORF72, la molecola esca ha inibito la tossicità.
“Abbiamo dimostrato, utilizzando molteplici approcci diversi, che la molecola che abbiamo progettato, “l’esca”, si lega in modo molto elevato e specifico a questa proteina poli-GR e, legandosi ad essa, le impedisce di legarsi all’RNA ribosomiale e le impedisce di entrare nel nucleo dove diventa più tossica“, ha spiegato Kiskinis. “Lo studio serve come prova di principio dell’idea che una molecola di RNA dotata di queste capacità di ‘esca’ può essere terapeutica e rafforza anche l’ipotesi che gran parte della tossicità derivante dalla mutazione C9ORF72 sia associata a un danno alla biologia ribosomiale“, aggiunge il ricercatore.
I ricercatori stanno ora ottimizzando la chimica della molecola esca e mirano a testarla in altri modelli di SLA.
“Siamo entusiasti del fatto che i neuroni derivati da iPSC di pazienti che presentano queste mutazioni sopravvivono molto più a lungo quando diamo loro questa molecola esca, il che significa che sta facendo qualcosa di buono. Dobbiamo solo capire se potrebbe essere un terapeutico vitale e trasformativo“, ha affermato Kiskinis.
Collegamento dei geni causali
Ci sono 30 geni conosciuti associati alle forme familiari di SLA, ma secondo Kiskinis, “se ciascun driver genetico contribuisca a un solo tipo generale di SLA o a più tipi rimane una questione incombente sul campo”.
Per rispondere a questa domanda, secondo lo studio pubblicato su Cell Reports, il team di Kiskinis ha utilizzato motoneuroni spinali derivati da cellule staminali pluripotenti indotte (iPSC) derivati dai pazienti per sviluppare un modello del secondo tipo più comune di SLA familiare, che è causata da mutazioni nel gene SOD1 ed è evidente nel 2% di tutti i casi di SLA.
“Sappiamo dal lavoro svolto sul campo negli ultimi 20 anni che questa mutazione causa tossicità attraverso effetti di guadagno di funzione. Sappiamo che le mutazioni portano al ripiegamento errato delle proteine e il ripiegamento errato avvia una cascata che porta a disfunzione. Ma come ciò accada non lo capiamo del tutto“, ha detto Kiskinis.
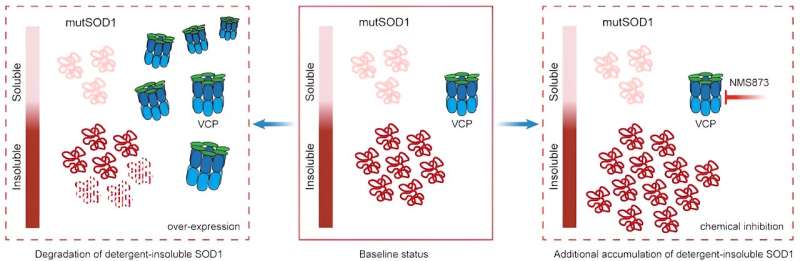
I ricercatori hanno utilizzato la proteomica temporale per confrontare i proteomi dei neuroni iPSC di controllo e dei neuroni iPSC con una mutazione SOD1 e hanno confrontato il tasso di degradazione delle proteine (il riciclo e la sostituzione delle vecchie proteine) tra entrambi i gruppi di neuroni.
Nei neuroni iPSC mutanti SOD1, hanno scoperto che un’altra proteina causale della SLA, “la proteina contenente valosina (VCP), si degrada più lentamente rispetto ai neuroni iPSC di controllo isogenico. Questa lenta degradazione proteica alla fine ha fatto sì che la VCP interagisse meno con alcune proteine e più con altre proteine”.
“Questo è piuttosto interessante perché si ritiene che le mutazioni nel VCP che possono causare la SLA agiscano in modo simile“, ha detto Kiskinis.
I ricercatori hanno quindi mirato a determinare se questa funzione alterata del VCP avesse un ruolo nella tossicità mediata da SDO1.Sia nei neuroni dei pazienti iPSC con SOD1 mutante che nei modelli di C. elegans (un tipo di nematode) di SOD1 mutante, i ricercatori hanno scoperto che quando VCP era sovraespresso, la tossicità diminuiva e quando la funzione VCP era inibita, la tossicità aumentava.
Leggi anche:SLA: nuovo composto si dimostra promettente per il trattamento
“Questo articolo e il lavoro svolto nel nostro laboratorio negli ultimi anni stanno dimostrando che, sebbene le cause genetiche possano essere diverse, c’è sempre una sorta di livello di sovrapposizione meccanicistica quando si tratta delle cause di disfunzione in queste cellule “, ha detto Kiskinis. “Questo è importante“, ha aggiunto, “perché i risultati possono informare la progettazione di futuri studi clinici e migliorare l’applicazione di terapie mirate“. Andando avanti, Kiskinis ha affermato che il suo team vuole determinare se “la VCP è un bersaglio terapeutico efficace per i pazienti con una mutazione SOD1, così come per altri tipi di SLA”.
Immagine Credit Public Domain-
Fonte:Science Advances