AlphaGenome-immagine Credit: Peter Kováč/Alamy
Ricercatori di un hackathon hanno utilizzato AlphaGenome per ricercare le cause genetiche di 29 malattie rare non diagnosticate.
Quando l’anno scorso più di 100 ricercatori si sono chiusi volontariamente in una stanza per affrontare alcune delle condizioni più difficili in campo medico, si sono rivolti all’intelligenza artificiale.
Nell’ambito di un’iniziativa, denominata Undiagnosed Hackathon, volta a individuare 29 patologie non diagnosticate, i ricercatori hanno utilizzato AlphaGenome, un modello di intelligenza artificiale sviluppato da Google DeepMind a Londra e descritto su Nature il 28 gennaio.
AlphaGenome è stato messo a disposizione degli scienziati lo scorso anno ed è in grado di prevedere i diversi effetti delle mutazioni nelle sequenze di DNA non codificanti, incluso il modo in cui potrebbero influenzare l’attività dei geni vicini.
Decifrare il 98% del genoma umano che non codifica proteine è una delle grandi sfide della biologia. Le mutazioni in queste sequenze sono particolarmente difficili per i ricercatori che cercano di scoprire le basi genetiche di malattie rare, spesso fatali.
“Si tratta di varianti che, a dire il vero, spesso vengono sottoposte a triage”, afferma Eric Klee, bioinformatico presso la Mayo Clinic di Rochester, Minnesota, che ha co-diretto l’Undiagnosed Hackathon nel settembre dello scorso anno.
Malattie rare non diagnosticate
L’evento di tre giorni presso la Mayo Clinic di Rochester, così come i due precedenti hackathon in Europa, sono stati organizzati dalla Wilhelm Foundation, un’organizzazione benefica di Brottby, in Svezia, che si occupa delle famiglie colpite da malattie rare non diagnosticate. L’organizzazione è stata fondata da Helene e Mikk Cederroth, che hanno perso tre dei loro quattro figli a causa di una malattia non diagnosticata e ha intitolato la fondazione al loro figlio maggiore, scomparso all’età di 16 anni.
Circa 350 milioni di persone soffrono di una malattia rara non diagnosticata, ma solo una piccola parte può essere diagnosticata utilizzando tecnologie esistenti come il sequenziamento del genoma. “Se non hai una diagnosi, sei lasciato indietro”, afferma Helene Cederroth.
Il lavoro sperimentale ha dimostrato che la mutazione alterava l’espressione genica nelle cellule cardiache, ma non in quelle neurali, il che era in linea con i sintomi manifestati dall’individuo. “Le previsioni di AlphaGenome sugli effetti della variante hanno supportato questa conclusione“, afferma Klee.
Il nuovo modello di deep learning di Google è in grado di prevedere l’effetto di piccole modifiche alle sequenze di DNA lunghe fino a un milione di coppie di basi ed è particolarmente efficace con il DNA non codificante, che si è dimostrato particolarmente difficile da comprendere. Lo strumento di intelligenza artificiale (IA), chiamato AlphaGenome, offre ai ricercatori un modo per comprendere meglio il genoma umano e potrebbe aiutare gli scienziati a sviluppare trattamenti per le malattie.
AlphaGenome è “uno strumento fondamentale e di alta qualità che trasforma il codice statico del genoma in un linguaggio decifrabile”, dice Robert Goldstone, Istituto Francis Crick.
Piccole variazioni nel genoma umano possono avere un impatto significativo sulla salute di una persona, causando malattie genetiche come la fibrosi cistica o alcuni tipi di cancro. La maggior parte dei cambiamenti si verifica nelle regioni non codificanti del genoma, che costituiscono il 98% del DNA totale. Queste regioni influenzano l’espressione dei geni, anziché codificare proteine e le alterazioni possono spesso avere una serie di effetti biologici, rendendo difficile prevederne l’impatto.
AlphaGenome, sviluppato da Google DeepMind, è in grado di prevedere l’impatto molecolare delle variazioni di singole coppie di basi su intere sequenze di DNA lunghe fino a un milione di coppie di basi. Questo si basa sul precedente modello di Google, AlphaMissense, che era in grado di comprendere solo gli effetti delle variazioni nella regione codificante delle sequenze di DNA.
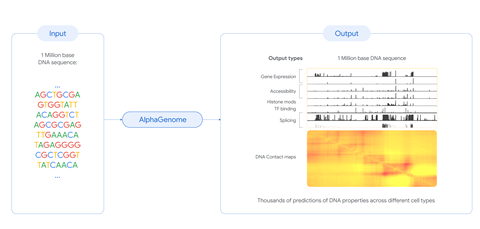
Il nuovo modello, basato su dati del genoma umano e murino, prende in input una sequenza di DNA e fornisce previsioni su vari segnali genetici correlati a specifiche funzioni biologiche, tra cui l’espressione genica, l’accessibilità del DNA alle proteine e la posizione dello splicing genico.
“Il vantaggio principale è che è possibile introdurre una mutazione nella sequenza, cambiando ad esempio una coppia di basi C in una T, e poi utilizzare il modello per confrontare queste differenze”, afferma Žiga Avsec, ricercatore di Google DeepMind.
Leggi anche:
Cosa intendiamo quando parliamo di IA?
L’intelligenza artificiale (IA) è un termine generico spesso utilizzato in modo errato per comprendere una varietà di processi interconnessi, ma più semplici.
L’intelligenza artificiale è la capacità delle macchine e dei programmi informatici di svolgere compiti che normalmente solo gli esseri umani potrebbero svolgere, come ragionare, rispondere al feedback e prendere decisioni.
L’intelligenza artificiale generativa è una variante più recente dell’intelligenza artificiale che analizza e rileva pattern nei set di dati di addestramento per generare testo, immagini e video originali in risposta alle richieste degli utenti. ChatGPT, Microsoft Copilot, Google Gemini e, più recentemente, Grok di X sono tutti esempi di chatbot che utilizzano l’intelligenza artificiale generativa.
Le reti neurali sono una serie interconnessa di neuroni artificiali, simili ai cervelli biologici, che identificano, analizzano e apprendono da modelli statistici nei dati.
Il machine learning è un sottoinsieme dell’intelligenza artificiale che consente alle macchine di apprendere dai set di dati e di fare previsioni basate su nuovi dati, senza che i programmatori lo richiedano esplicitamente. I modelli di machine learning migliorano le loro prestazioni man mano che ricevono più dati.
Il deep learning è una tipologia avanzata di apprendimento automatico che utilizza reti neurali a più livelli per analizzare dati complessi provenienti da set di dati molto ampi. Le applicazioni del deep learning includono il riconoscimento vocale, la generazione di immagini e la traduzione.
I modelli linguistici di grandi dimensioni (LLM) sono un tipo di apprendimento profondo addestrato su grandi quantità di dati per comprendere e generare il linguaggio. Gli LLM apprendono gli schemi nel testo prevedendo la parola successiva nella sequenza e sono ora in grado di scrivere prosa, analizzare testi da Internet e dialogare con gli utenti.
AlphaGenome ha eguagliato o superato altri modelli all’avanguardia in 25 dei 26 compiti di previsione degli effetti delle variazioni genetiche. Il team è stato anche in grado di simulare mutazioni note del DNA responsabili di un tipo di leucemia, prevedendo gli stessi risultati osservati in laboratorio.
“In precedenza, il settore richiedeva modelli separati per compiti separati“, afferma Avsec, aggiungendo che i modelli precedenti spesso richiedevano anche un compromesso tra lunghezza della sequenza e risoluzione. “AlphaGenome li unifica sotto lo stesso tetto”. Natasha Latysheva, ingegnere di ricerca senior presso DeepMind, spiega che AlphaGenome potrebbe contribuire a migliorare le conoscenze fondamentali sul genoma, a migliorare la comprensione delle malattie rare e dei tumori o ad aiutare gli scienziati a progettare nuove sequenze di DNA per curare condizioni specifiche.
AlphaGenome si aggiunge alla collezione di altri strumenti di intelligenza artificiale sviluppati da Google DeepMind, che include AlphaFold, vincitore del premio Nobel 2024, che prevede la forma tridimensionale delle proteine. Pushmeet Kohli, che ha guidato il lavoro, spiega che “il genoma è la ricetta e comprendere l’effetto di una qualsiasi modifica di questa ricetta è ciò che AlphaGenome si propone di fare“.
AlphaGenome trasforma il codice genetico in un “linguaggio decifrabile di scoperta”
Robert Goldstone, responsabile della genomica presso il Francis Crick Institute nel Regno Unito, ritiene che AlphaGenome sia “uno strumento fondamentale e di alta qualità che trasforma il codice statico del genoma in un linguaggio decifrabile per la scoperta“, ma avverte che “non è una soluzione magica per tutte le questioni biologiche”.
Nonostante i miglioramenti, AlphaGenome presenta ancora una serie di limitazioni. Come altri modelli, fatica a prevedere l’influenza di alterazioni genetiche distanti più di 100.000 coppie di basi e può fare previsioni solo sulle sequenze di DNA dei tipi cellulari utilizzati per addestrare il modello, ovvero umani e topi.
“Un altro problema è l’interpretazione dei risultati del modello”, spiega Jian Zhou, ricercatore di apprendimento automatico in genomica presso l’Università di Chicago negli Stati Uniti. “Anche quando il modello fa previsioni accurate, non sempre ci informa direttamente sui processi biologici sottostanti“, aggiunge.
Google DeepMind ha pubblicato un’anteprima del modello per la ricerca non commerciale a giugno dello scorso anno. Da allora, Kohli spiega che quasi 3000 scienziati in 160 paesi diversi hanno utilizzato AlphaGenome, inviando circa 1 milione di richieste al giorno.
Spera che “AlphaGenome continuerà a essere una risorsa preziosa per la comunità scientifica e ad aiutare gli scienziati a comprendere meglio la funzione del genoma e la biologia delle malattie, e in ultima analisi a promuovere nuove scoperte biologiche e… nuovi trattamenti”.
Fonte:Nature

