SLA-immagine: identificata la firma trascrizionale negli assoni motori e non motori del nervo sciatico maturo mediante trascrittomica spaziale. Crediti: Nature Neuroscience
Il funzionamento dei neuroni, le cellule che trasmettono le informazioni nel sistema nervoso, dipende in larga misura dalla produzione di proteine. Le proteine vengono sintetizzate sia all’interno delle cellule (ovvero nel soma) sia localmente lungo gli assoni, proiezioni attraverso le quali i segnali vengono trasmessi ad altre cellule.
La sintesi locale di proteine consente agli assoni di rispondere rapidamente ai cambiamenti, crescere e ripararsi, il che a sua volta consente la formazione di nuove connessioni nel cervello associate all’apprendimento e alla memoria. Studi precedenti hanno collegato le interruzioni nella produzione locale di proteine ad alcune malattie neurodegenerative, tra cui la sclerosi laterale amiotrofica (SLA).
La SLA è caratterizzata dalla progressiva degradazione dei motoneuroni, le cellule nervose che supportano i movimenti muscolari volontari. I ricercatori del Leuven Brain Institute e della KU Leuven in Belgio hanno raccolto nuove preziose informazioni sul contributo della sintesi proteica locale a questa grave malattia neurodegenerativa, pubblicate su Nature Neuroscience.
Studio dei neuroni portatori di mutazioni FUS legate alla SLA
Per esplorare il legame tra la sintesi proteica negli assoni e la SLA, il team ha analizzato i motoneuroni dei topi utilizzando una tecnica nota come trascrittomica spaziale. La trascrittomica spaziale consente ai neuroscienziati di mappare l’espressione genica direttamente all’interno di sezioni di tessuto, gettando nuova luce su come specifici tipi di cellule e la loro posizione supportino specifiche funzioni biologiche.

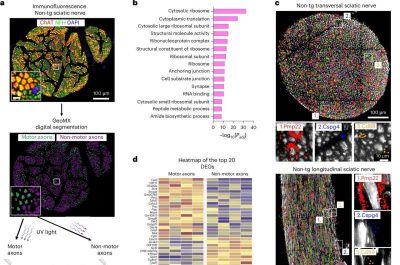
Immagine: Schema sperimentale del flusso di lavoro applicato per identificare trascrizioni specifiche dei motoneuroni utilizzando sonde di RNA fotoclivabili con luce UV contro l’intero genoma di topo in combinazione con anticorpi specifici per le cellule, utilizzando la piattaforma di trascrittomica spaziale NanoString GeoMx DSP. In alto, immagine rappresentativa di immunofluorescenza di una sezione di midollo spinale di topo utilizzando anticorpi ChAT e NFH per identificare i corpi cellulari dei motoneuroni (riquadro a sinistra) e dei neuroni non motori (riquadro a destra). Al centro, immagine rappresentativa della segmentazione digitale applicata utilizzando il software GeoMX DSP. In basso, sequenziamento di nuova generazione di RNA rilasciati con luce laser UV estratti da neuroni motori o neuroni non motori raggruppati. Barre di scala, 500 µm e 50 µm (riquadro). Crediti: Nature
Utilizzando la trascrittomica spaziale, Piol, Khalil e i loro colleghi hanno identificato i trascritti di RNA più ricchi negli assoni o nei somi cellulari. Hanno poi utilizzato un’altra tecnica chiamata immunofluorescenza per visualizzare le proteine negli assoni e nelle cellule del topo, rilevando l’RNA che supporta la sintesi proteica. Nelle loro analisi, il team si è concentrato sulle mutazioni di una proteina specifica, chiamata FUS (fused in sarcoma), che si lega alle molecole di RNA e ne regola la stabilità, il trasporto e la traduzione.
“Tra le trascrizioni trovate negli assoni maturi, il processo biologico più arricchito è la traduzione proteica, e la localizzazione del meccanismo di traduzione è stata confermata utilizzando la trascrittomica spaziale a singola molecola multiplexata combinata con l’immunofluorescenza“, hanno scritto gli autori.
“Le mutazioni associate alla sclerosi laterale amiotrofica (SLA) nella proteina legante l’RNA fusa nel sarcoma (FUS), che sopprimono la traduzione locale, interrompono le firme di RNA specifiche del compartimento, inclusi i componenti del meccanismo di traduzione. In particolare, il fattore di inizio eucariotico 5a (Eif5a), un fattore di traduzione coinvolto nell’allungamento e nella terminazione, è localmente compromesso negli assoni FUS mutanti con livelli ridotti della sua forma ipusinata attiva“.
Guidare lo sviluppo delle future terapie per la SLA
Piol, Khalil e i loro colleghi hanno trovato prove che suggeriscono che le proteine sono parzialmente sintetizzate all’interno degli assoni, ma questa produzione locale di proteine viene interrotta quando i neuroni presentano mutazioni nel gene FUS. Queste mutazioni FUS portano a una ridotta produzione di proteine nelle aree in cui sono più necessarie, un fenomeno precedentemente associato alla SLA.
Nell’ambito del loro studio, i ricercatori hanno anche cercato di ripristinare la sintesi proteica locale nei modelli di SLA nei moscerini della frutta, utilizzando una molecola naturale chiamata spermidina. Hanno scoperto che il trattamento dei topi con questa molecola ripristinava la traduzione proteica negli assoni, riducendo il danno ai neuroni.
Leggi anche:SLA: identificato un nuovo bersaglio terapeutico
“Il trattamento assonale specifico con poliammina spermidina ripristina l’ipusinazione di Eif5a e migliora i difetti neuronali dipendenti da FUS mutante, inclusa la soppressione della sintesi proteica locale”, hanno scritto Piol, Khalil e colleghi. “Infine, il trattamento in vivo con spermidina riduce la tossicità correlata alla SLA nei modelli mutanti di FUS e TDP-43 in Drosophila, il che potrebbe avere implicazioni per lo sviluppo di terapie“.
Studi futuri potrebbero tentare di replicare queste recenti scoperte in ulteriori esperimenti con altri modelli animali di SLA, inclusi i modelli murini. Infine, se convalidate nell’uomo, le conoscenze acquisite da questo team di ricerca potrebbero orientare lo sviluppo di nuove strategie terapeutiche che mitigano la neurodegenerazione ripristinando i processi di sintesi proteica locale.
Fonte: Nature Neuroscience